Durante la reazione PCR si incontrano spesso alcuni fattori interferenti.
Data l'altissima sensibilità della PCR, la contaminazione è considerata uno dei fattori più importanti che influenzano i risultati della PCR e può produrre risultati falsi positivi.
Altrettanto critiche sono le varie fonti che portano a risultati falsi negativi. Se una o più componenti essenziali della miscela di PCR o la reazione di amplificazione stessa vengono inibite o interferite, il test diagnostico può essere compromesso. Ciò può portare a una riduzione dell'efficienza e persino a risultati falsi negativi.
Oltre all'inibizione, la perdita di integrità dell'acido nucleico bersaglio può verificarsi a causa delle condizioni di trasporto e/o conservazione prima della preparazione del campione. In particolare, temperature elevate o una conservazione inadeguata possono danneggiare le cellule e gli acidi nucleici. La fissazione di cellule e tessuti e l'inclusione in paraffina sono cause ben note di frammentazione del DNA e rappresentano un problema persistente (vedere Figure 1 e 2). In questi casi, anche l'isolamento e la purificazione ottimali non saranno d'aiuto.
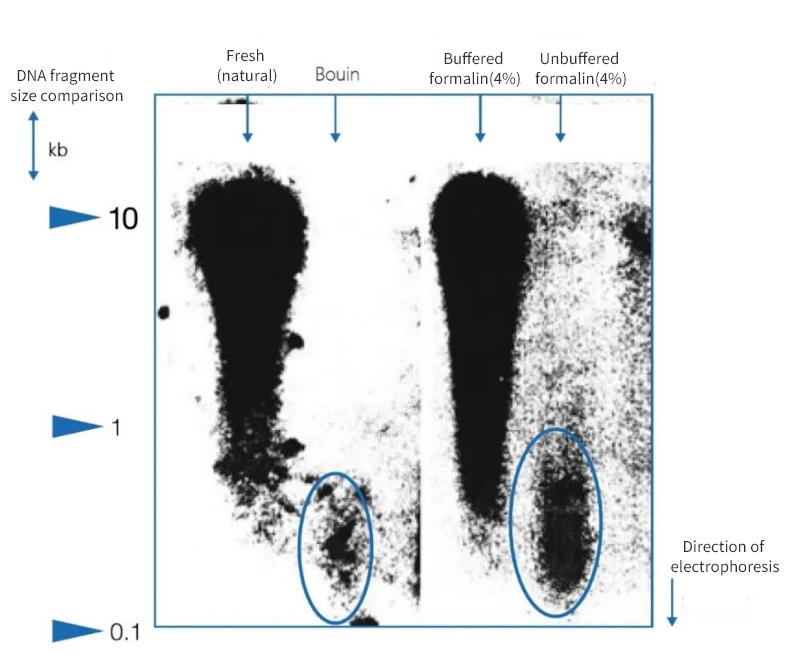
Figura 1 | Effetto dell'immobilizzazione sull'integrità del DNA
L'elettroforesi su gel di agarosio ha mostrato che la qualità del DNA isolato dalle sezioni di paraffina delle autopsie variava considerevolmente. Negli estratti era presente DNA con frammenti di lunghezza media diversa a seconda del metodo di fissazione. Il DNA è stato preservato solo se fissato in campioni congelati nativi e in formalina neutra tamponata. L'uso di un fissativo Bouin fortemente acido o di formalina non tamponata contenente acido formico ha comportato una perdita significativa di DNA. La frazione rimanente è altamente frammentata.
A sinistra, la lunghezza dei frammenti è espressa in coppie di kilobase (kbp)
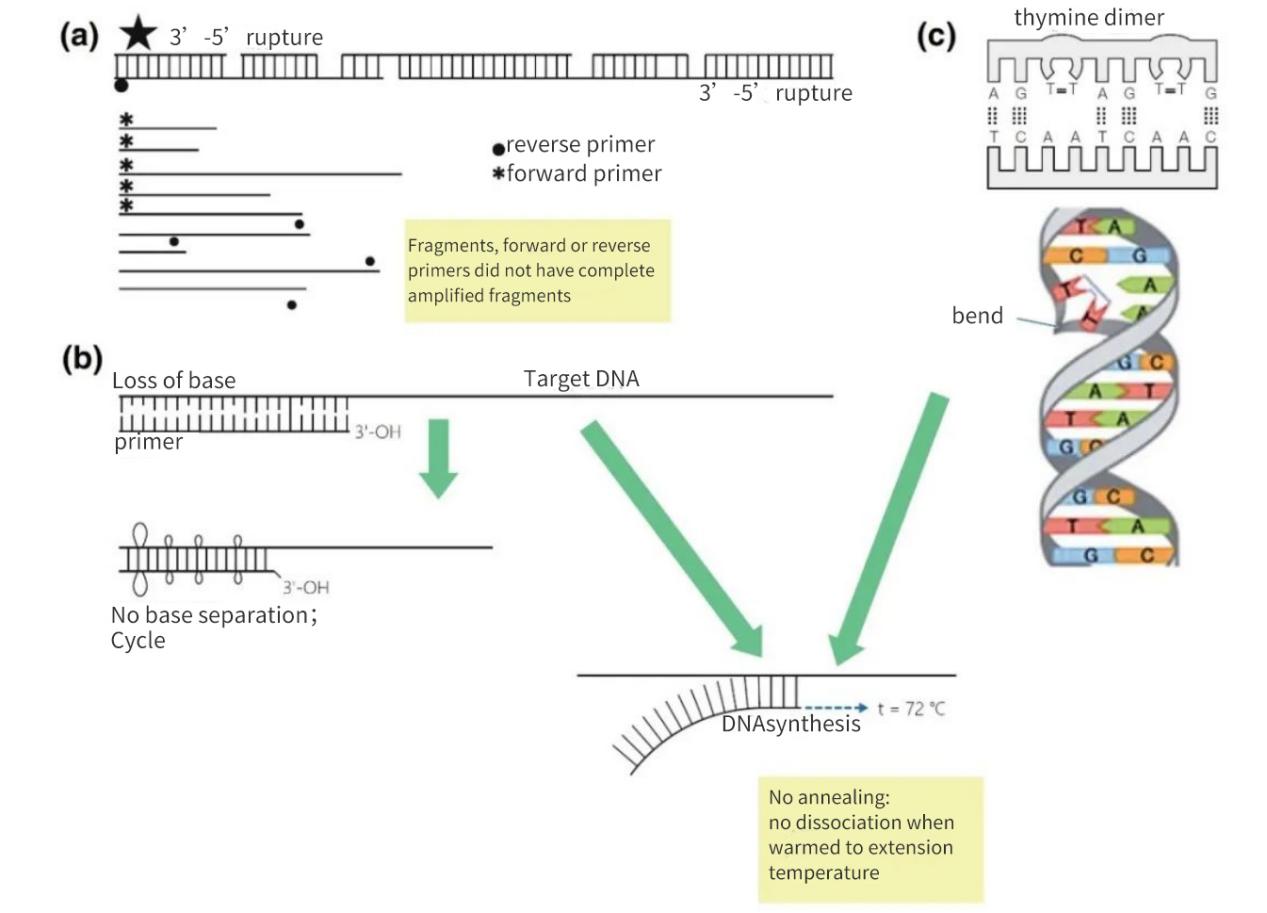
Figura 2 | Perdita di integrità dei bersagli degli acidi nucleici
(a) Un gap 3'-5' su entrambi i filamenti provocherà una rottura nel DNA bersaglio. La sintesi del DNA avverrà comunque sul piccolo frammento. Tuttavia, se un sito di annealing del primer è mancante sul frammento di DNA, si verifica solo un'amplificazione lineare. Nel caso più favorevole, i frammenti potrebbero risaturarsi a vicenda, ma le rese saranno ridotte e al di sotto dei livelli di rilevamento.
(b) La perdita di basi, dovuta principalmente alla depurinazione e alla formazione di dimeri di timidina, porta a una diminuzione del numero di legami a idrogeno e a una diminuzione della Tm. Durante la fase di riscaldamento prolungata, i primer si scioglieranno dal DNA della matrice e non si ricotturano nemmeno in condizioni meno stringenti.
(c) Le basi di timina adiacenti formano un dimero TT.
Un altro problema comune che si verifica spesso nella diagnostica molecolare è il rilascio non ottimale degli acidi nucleici target rispetto all'estrazione con fenolo-cloroformio. In casi estremi, questo può essere associato a falsi negativi. Si può risparmiare molto tempo con la lisi per bollitura o la digestione enzimatica dei detriti cellulari, ma questo metodo spesso si traduce in una bassa sensibilità della PCR a causa dell'insufficiente rilascio di acidi nucleici.
Inibizione dell'attività della polimerasi durante l'amplificazione
In generale, l'inibizione è utilizzata come concetto contenitore per descrivere tutti i fattori che portano a risultati PCR subottimali. In senso strettamente biochimico, l'inibizione è limitata all'attività dell'enzima, ovvero riduce o impedisce la conversione substrato-prodotto attraverso l'interazione con il sito attivo della DNA polimerasi o con il suo cofattore (ad esempio, Mg2+ per la Taq DNA polimerasi).
I componenti del campione o vari tamponi ed estratti contenenti reagenti possono inibire direttamente l'enzima o intrappolare i suoi cofattori (ad esempio EDTA), inattivando così la polimerasi e determinando a sua volta risultati PCR ridotti o falsi negativi.
Tuttavia, molte interazioni tra i componenti della reazione e gli acidi nucleici contenenti il bersaglio sono anche designate come "inibitori della PCR". Una volta che l'integrità della cellula viene compromessa dall'isolamento e l'acido nucleico viene rilasciato, possono verificarsi interazioni tra il campione e la soluzione circostante e la fase solida. Ad esempio, gli "scavenger" possono legare il DNA a singolo o doppio filamento attraverso interazioni non covalenti e interferire con l'isolamento e la purificazione riducendo il numero di bersagli che alla fine raggiungono il contenitore di reazione della PCR.
In generale, gli inibitori della PCR sono presenti nella maggior parte dei fluidi corporei e dei reagenti utilizzati per i test diagnostici clinici (urea nelle urine, emoglobina ed eparina nel sangue), negli integratori alimentari (componenti organici, glicogeno, grassi, ioni Ca2+) e nei componenti ambientali (fenoli, metalli pesanti).
| Inibitori | Fonte |
| ioni calcio | Latte, tessuto osseo |
| collagene | Tessuto |
| Sali biliari | Feci |
| Emoglobina | Nel sangue |
| Emoglobina | campioni di sangue |
| Acido umico | Suolo, pianta |
| Sangue | Sangue |
| Lattoferrina | Sangue |
| melanina (europea) | Pelle, capelli |
| Mioglobina | Tessuto muscolare |
| Polisaccaridi | Pianta, feci |
| Proteasi | Latte |
| Urea | urina |
| Mucopolisaccaride | Cartilagine, mucose |
| Lignina, cellulosa | Piante |
Gli inibitori della PCR più diffusi si trovano nei batteri e nelle cellule eucariotiche, nel DNA non bersaglio, nelle macromolecole che legano il DNA delle matrici tissutali e nelle attrezzature di laboratorio come guanti e plastica. La purificazione degli acidi nucleici durante o dopo l'estrazione è il metodo preferito per rimuovere gli inibitori della PCR.
Oggi, diverse apparecchiature di estrazione automatizzate possono sostituire molti protocolli manuali, ma non è mai stato raggiunto il 100% di recupero e/o purificazione dei target. Potenziali inibitori potrebbero essere ancora presenti negli acidi nucleici purificati o potrebbero aver già fatto effetto. Esistono diverse strategie per ridurre l'impatto degli inibitori. La scelta della polimerasi appropriata può avere un impatto significativo sull'attività dell'inibitore. Altri metodi comprovati per ridurre l'inibizione della PCR consistono nell'aumentare la concentrazione della polimerasi o nell'applicare additivi come la BSA.
L'inibizione delle reazioni PCR può essere dimostrata mediante l'uso del controllo di qualità del processo interno (IPC).
È necessario prestare attenzione a rimuovere tutti i reagenti e le altre soluzioni presenti nel kit di estrazione, come etanolo, EDTA, CETAB, LiCl, GuSCN, SDS, isopropanolo e fenolo, dall'acido nucleico isolato mediante un'accurata fase di lavaggio. A seconda della loro concentrazione, possono attivare o inibire la PCR.
Data di pubblicazione: 19-05-2023
 中文网站
中文网站