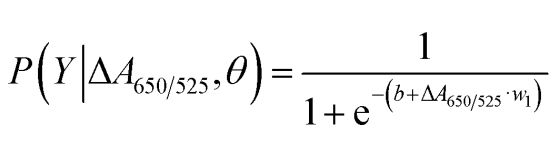La diagnosi precoce del cancro basata sulla biopsia liquida è una nuova direzione nella diagnosi e nella diagnosi del cancro proposta dal National Cancer Institute degli Stati Uniti negli ultimi anni, con l'obiettivo di individuare tumori in fase iniziale o addirittura lesioni precancerose. È stata ampiamente utilizzata come nuovo biomarcatore per la diagnosi precoce di diverse neoplasie, tra cui cancro ai polmoni, tumori gastrointestinali, gliomi e tumori ginecologici.
L'emergere di piattaforme per identificare i biomarcatori del panorama di metilazione (Methylscape) ha il potenziale di migliorare significativamente gli attuali screening precoci per il cancro, consentendo ai pazienti di raggiungere la fase più precoce possibile del trattamento.
Recentemente, i ricercatori hanno sviluppato una piattaforma di rilevamento semplice e diretta per il rilevamento del paesaggio di metilazione basata su nanoparticelle d'oro decorate con cisteamina (Cyst/AuNP) combinate con un biosensore per smartphone che consente uno screening rapido e precoce di un'ampia gamma di tumori. Lo screening precoce per la leucemia può essere eseguito entro 15 minuti dall'estrazione del DNA da un campione di sangue, con un'accuratezza del 90,0%. Il titolo dell'articolo è "Rilevamento rapido del DNA tumorale nel sangue umano utilizzando AuNP ricoperte di cisteamina e uno smartphone con apprendimento automatico".
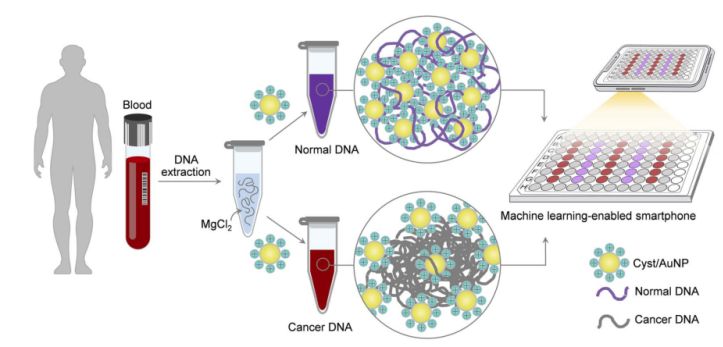
Figura 1. Una piattaforma di rilevamento semplice e veloce per lo screening del cancro tramite componenti Cyst/AuNP può essere realizzata in due semplici passaggi.
Ciò è mostrato nella Figura 1. Innanzitutto, è stata utilizzata una soluzione acquosa per dissolvere i frammenti di DNA. Alla soluzione mista sono state poi aggiunte cisti/AuNP. Il DNA normale e quello maligno presentano diverse proprietà di metilazione, che danno origine a frammenti di DNA con diversi schemi di autoassemblaggio. Il DNA normale si aggrega debolmente e alla fine aggrega cisti/AuNP, il che determina la natura redshift di queste ultime, tanto che un cambiamento di colore dal rosso al viola può essere osservato a occhio nudo. Al contrario, il profilo di metilazione unico del DNA tumorale porta alla produzione di cluster più grandi di frammenti di DNA.
Le immagini delle piastre a 96 pozzetti sono state acquisite utilizzando la fotocamera di uno smartphone. Il DNA tumorale è stato misurato tramite uno smartphone dotato di apprendimento automatico, rispetto ai metodi basati sulla spettroscopia.
Screening del cancro su campioni di sangue reali
Per ampliare l'utilità della piattaforma di rilevamento, i ricercatori hanno utilizzato un sensore in grado di distinguere con successo il DNA normale da quello canceroso in campioni di sangue reali. I pattern di metilazione nei siti CpG regolano epigeneticamente l'espressione genica. In quasi tutti i tipi di cancro, è stata osservata un'alternanza di alterazioni nella metilazione del DNA e quindi nell'espressione dei geni che promuovono la tumorigenesi.
Come modello per altri tumori associati alla metilazione del DNA, i ricercatori hanno utilizzato campioni di sangue di pazienti affetti da leucemia e di controlli sani per studiare l'efficacia del panorama di metilazione nella differenziazione dei tumori leucemici. Questo biomarcatore del panorama di metilazione non solo supera i metodi di screening rapido della leucemia esistenti, ma dimostra anche la fattibilità di estenderlo alla diagnosi precoce di un'ampia gamma di tumori utilizzando questo test semplice e diretto.
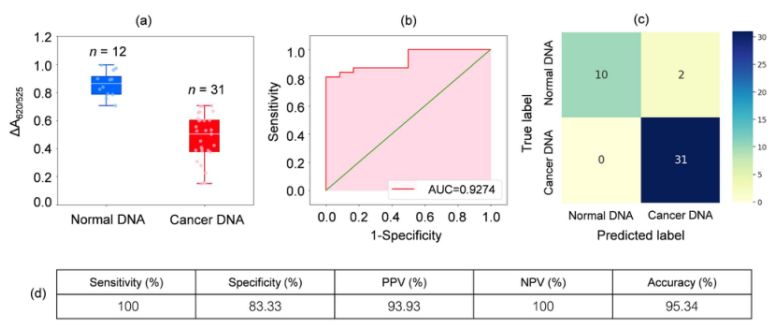
È stato analizzato il DNA da campioni di sangue di 31 pazienti affetti da leucemia e 12 individui sani. Come mostrato nel box plot in Figura 2a, l'assorbanza relativa dei campioni tumorali (ΔA650/525) era inferiore a quella del DNA di campioni normali. Ciò era dovuto principalmente all'aumentata idrofobicità che portava a una densa aggregazione del DNA tumorale, impedendo l'aggregazione di cisti/AuNP. Di conseguenza, queste nanoparticelle erano completamente disperse negli strati esterni degli aggregati tumorali, il che determinava una diversa dispersione di cisti/AuNP adsorbite sugli aggregati di DNA normale e tumorale. Le curve ROC sono state quindi generate variando la soglia da un valore minimo di ΔA650/525 a un valore massimo.
Figura 2.(a) Valori di assorbanza relativa delle soluzioni di cisti/AuNP che mostrano la presenza di DNA normale (blu) e canceroso (rosso) in condizioni ottimizzate
(DA650/525) dei box plot; (b) Analisi ROC e valutazione dei test diagnostici. (c) Matrice di confusione per la diagnosi di pazienti normali e oncologici. (d) Sensibilità, specificità, valore predittivo positivo (VPP), valore predittivo negativo (VPN) e accuratezza del metodo sviluppato.
Come mostrato in Figura 2b, l'area sotto la curva ROC (AUC = 0,9274) ottenuta per il sensore sviluppato ha mostrato elevata sensibilità e specificità. Come si può osservare dal box plot, il punto più basso che rappresenta il gruppo di DNA normale non è ben separato dal punto più alto che rappresenta il gruppo di DNA tumorale; pertanto, è stata utilizzata la regressione logistica per differenziare i gruppi di DNA normale da quelli tumorali. Dato un insieme di variabili indipendenti, questa stima la probabilità che si verifichi un evento, come un gruppo di DNA tumorale o normale. La variabile dipendente varia tra 0 e 1. Il risultato è quindi una probabilità. Abbiamo determinato la probabilità di identificazione del cancro (P) in base a ΔA650/525 come segue.
dove b=5,3533, w1=-6,965. Per la classificazione del campione, una probabilità inferiore a 0,5 indica un campione normale, mentre una probabilità pari o superiore a 0,5 indica un campione canceroso. La Figura 2c illustra la matrice di confusione generata dalla convalida incrociata "leave-it-alone", utilizzata per convalidare la stabilità del metodo di classificazione. La Figura 2d riassume la valutazione del test diagnostico del metodo, inclusi sensibilità, specificità, valore predittivo positivo (VPP) e valore predittivo negativo (VPN).
Biosensori basati su smartphone
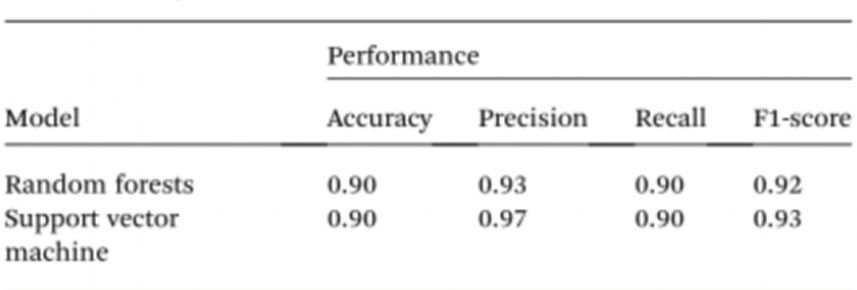
Per semplificare ulteriormente l'analisi dei campioni senza l'uso di spettrofotometri, i ricercatori hanno utilizzato l'intelligenza artificiale (IA) per interpretare il colore della soluzione e distinguere tra individui normali e cancerosi. A tale scopo, è stata utilizzata la visione artificiale per tradurre il colore della soluzione di cisti/AuNP in DNA normale (viola) o DNA canceroso (rosso) utilizzando immagini di piastre a 96 pozzetti acquisite tramite la fotocamera di un telefono cellulare. L'intelligenza artificiale può ridurre i costi e migliorare l'accessibilità nell'interpretazione del colore delle soluzioni di nanoparticelle, senza l'uso di accessori ottici per smartphone. Infine, due modelli di apprendimento automatico, tra cui Random Forest (RF) e Support Vector Machine (SVM), sono stati addestrati per costruire i modelli. Entrambi i modelli RF e SVM hanno classificato correttamente i campioni come positivi e negativi con un'accuratezza del 90,0%. Ciò suggerisce che l'uso dell'intelligenza artificiale nella biosensoristica basata su telefoni cellulari è del tutto possibile.
Figura 3.(a) Classe target della soluzione registrata durante la preparazione del campione per la fase di acquisizione dell'immagine. (b) Immagine di esempio acquisita durante la fase di acquisizione dell'immagine. (c) Intensità del colore della soluzione cisti/AuNP in ciascun pozzetto della piastra a 96 pozzetti estratta dall'immagine (b).
Utilizzando Cyst/AuNP, i ricercatori hanno sviluppato con successo una semplice piattaforma di rilevamento per il rilevamento del paesaggio di metilazione e un sensore in grado di distinguere il DNA normale da quello tumorale quando si utilizzano campioni di sangue reale per lo screening della leucemia. Il sensore sviluppato ha dimostrato che il DNA estratto da campioni di sangue reale era in grado di rilevare rapidamente e a basso costo piccole quantità di DNA tumorale (3nM) in pazienti affetti da leucemia in 15 minuti, con un'accuratezza del 95,3%. Per semplificare ulteriormente l'analisi dei campioni eliminando la necessità di uno spettrofotometro, è stato utilizzato l'apprendimento automatico per interpretare il colore della soluzione e distinguere tra individui normali e tumorali utilizzando una fotografia scattata con un telefono cellulare, raggiungendo un'accuratezza del 90,0%.
Riferimento: DOI: 10.1039/d2ra05725e
Data di pubblicazione: 18-02-2023
 中文网站
中文网站